
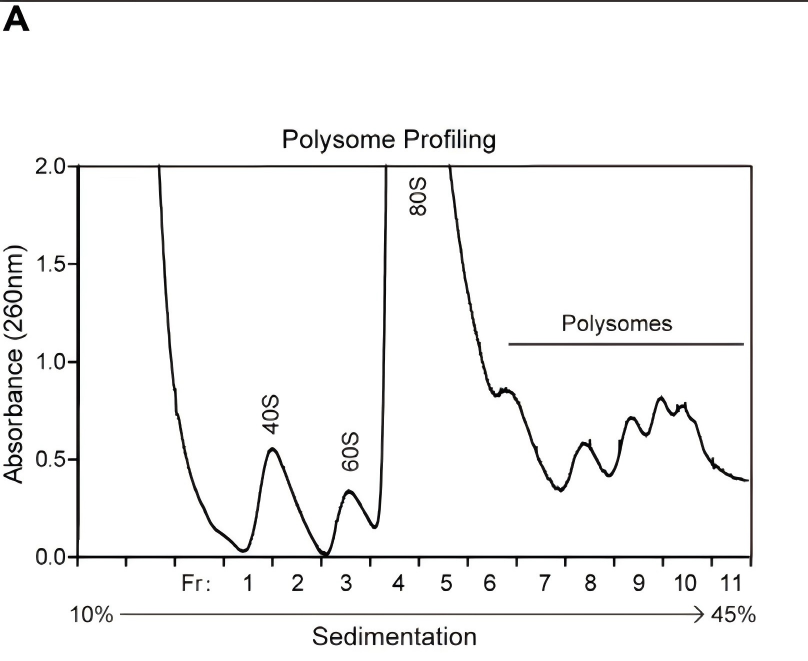
Polysome profiling是基于蔗糖密度梯度離心,將細(xì)胞質(zhì)RNA分離成多個(gè)組分:游離RNA,結(jié)合核糖體40S或60S亞基、單核糖體(80S)或多聚核糖體等。后續(xù)可以分別收集40S、60S、80S和多聚核糖體,用于RNA或蛋白質(zhì)分析,也可以通過(guò)酶消化檢測(cè)disome等等。
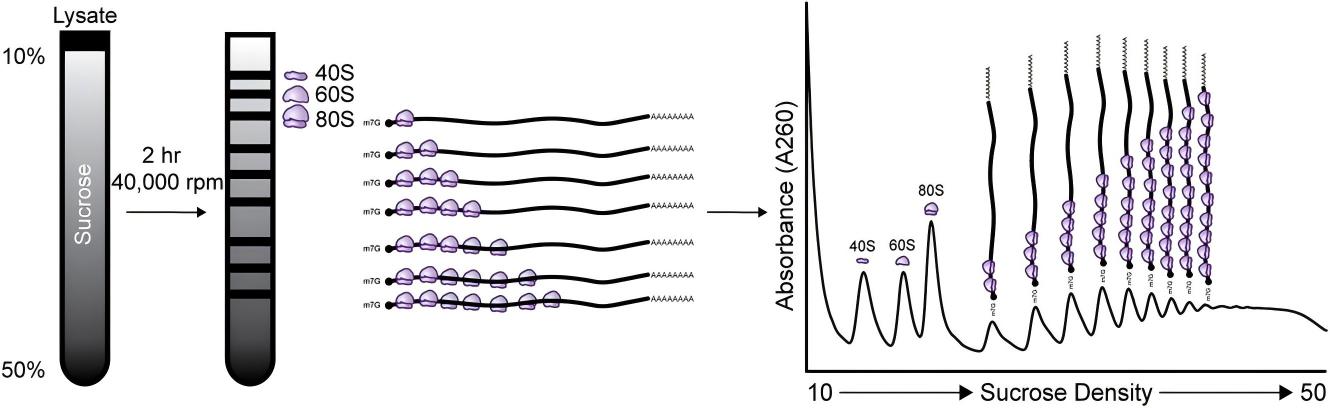
1:Disome/Trisome-seq
在polysome profiling基礎(chǔ)上進(jìn)行酶切處理,酶切去除未被核糖體保護(hù)的mRNA,只測(cè)序核糖體保護(hù)的短片段(RPF);通過(guò)發(fā)現(xiàn)disome或trisome的變化來(lái)檢測(cè)核糖體碰撞(collision)與翻譯停滯(stalling)現(xiàn)象。
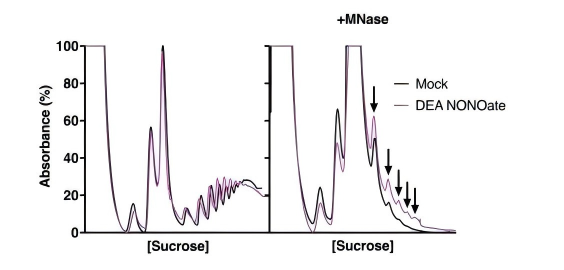
文獻(xiàn)案例:突變亨廷頓蛋白在亨廷頓病細(xì)胞模型中阻滯核糖體并抑制蛋白質(zhì)合成

文章題目:Mutant Huntingtin stalls ribosomes and represses protein synthesis in a cellular model of Huntington disease
這篇研究論文主要探討了亨廷頓病(Huntington's disease, HD)中突變亨廷頓蛋白(mutant huntingtin, mHTT)如何通過(guò)影響核糖體功能和蛋白質(zhì)合成來(lái)導(dǎo)致神經(jīng)退行性病變的機(jī)制,為理解HD的發(fā)病機(jī)制和開(kāi)發(fā)治療方法提供了重要線索。
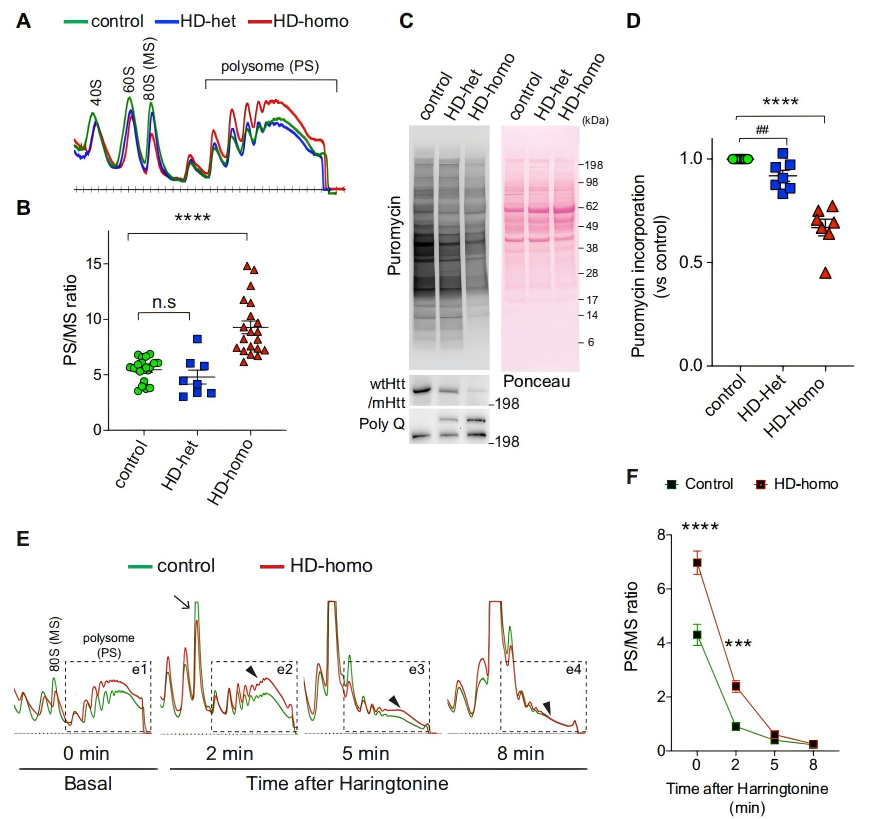
Hectd1 HD 細(xì)胞蛋白質(zhì)合成抑制和核糖體停滯
文獻(xiàn)案例:Disome-seq揭示了促進(jìn)共翻譯蛋白折疊的廣泛核糖體碰撞成

文章題目:Disome-seq reveals widespread ribosome collisions that promote cotranslational protein folding
研究通過(guò)創(chuàng)新的 Disome-seq 技術(shù)(測(cè)序雙核糖體保護(hù)的 mRNA 片段),系統(tǒng)揭示了酵母細(xì)胞中廣泛存在的核糖體碰撞現(xiàn)象及其在蛋白質(zhì)折疊調(diào)控中的關(guān)鍵作用。該研究證明核糖體碰撞是細(xì)胞調(diào)控蛋白質(zhì)穩(wěn)態(tài)的普遍策略,通過(guò)序列特異的翻譯暫停和伴侶蛋白協(xié)同,確保新生肽鏈的正確折疊,為理解蛋白質(zhì)折疊疾病(如神經(jīng)退行性疾病)提供了新視角。

Disome-seq 測(cè)定核糖體碰撞
2:Polysome-seq 多聚核糖體結(jié)合轉(zhuǎn)錄本測(cè)序
在polysome profiling的基礎(chǔ)上,提取多聚核糖體各組分樣本中的RNA,進(jìn)行RNA-seq測(cè)序,深入分析特定mRNA的翻譯狀態(tài)和多聚核糖體分布特征。


文獻(xiàn)案例:RNA 結(jié)合蛋白 PRRC2B 介導(dǎo)特定 mRNA 的翻譯并調(diào)節(jié)細(xì)胞周期進(jìn)程

文章題目:RNA binding protein PRRC2B mediates translation of specific mRNAs and regulates cell cycle progression
PRRC2B 通過(guò)結(jié)合特定 mRNA 的 5'UTR 區(qū),招募 eIF4G2/eIF3 復(fù)合物促進(jìn)翻譯,進(jìn)而驅(qū)動(dòng)細(xì)胞周期進(jìn)程。技術(shù)借鑒:整合 PAR-CLIP、多核糖體測(cè)序、質(zhì)譜互作組分析等技術(shù)。該研究揭示了 RNA 結(jié)合蛋白在翻譯調(diào)控中的新機(jī)制,為癌癥治療提供了潛在靶點(diǎn)。

PRRC2B 結(jié)合的 mRNA 在 PRRC2B 敲除后表現(xiàn)出翻譯效率降低
注:A 圖為 Polysome profiling 流程示意圖,D 和 E 圖為 Polysome-seq 分析翻譯效率差異圖,F(xiàn) 和 G 圖為 Polysome-qPCR 分析特定基因翻譯效率圖。
3:Polysome profiling+RT-qPCR
梯度分離多聚核糖體區(qū)域后,用RT-qPCR定量特定基因在各組分的分布,可用于目標(biāo)基因翻譯活躍度驗(yàn)證,藥物或處理?xiàng)l件下翻譯變化監(jiān)測(cè)
[內(nèi)參法}文獻(xiàn)案例:利用多聚核糖體譜分析和定量PCR鑒定懸浮細(xì)胞系中潛在的微肽編碼長(zhǎng)鏈非編碼RNA

文章題目:Polysome profiling followed by quantitative PCR for identifying potential micropeptide encoding long non-coding RNAs in suspension cell lines
該研究是通過(guò)懸浮細(xì)胞系中的多核糖體分析來(lái)篩選潛在的微肽編碼lncRNA。當(dāng)與定量PCR相結(jié)合,有助于同時(shí)識(shí)別許多翻譯lncRNA。
作者在完成polysome分離后,對(duì)各組分進(jìn)行了RNA提取和cDNA反轉(zhuǎn)錄。在通過(guò) RT-qPCR篩選可能翻譯的lncRNA時(shí),設(shè)置了陽(yáng)性和陰性對(duì)照,以評(píng)價(jià)結(jié)果的穩(wěn)健性。GAPDH等管家基因的mRNA可作為陽(yáng)性對(duì)照;已知的細(xì)胞質(zhì)非編碼RNA,如hY1,可以作為陰性對(duì)照。GAPDH的峰值應(yīng)位于高多核組分中,而hY1應(yīng)富集在亞單體組分中。在 polysome組分中具有突出峰值的候選lncRNA可能表明在細(xì)胞中激活翻譯。
qPCR數(shù)據(jù)分析
每個(gè)組分中RNA百分比的計(jì)算如下:

X = 計(jì)算的組分?jǐn)?shù);Y= 組分?jǐn)?shù)總數(shù)。
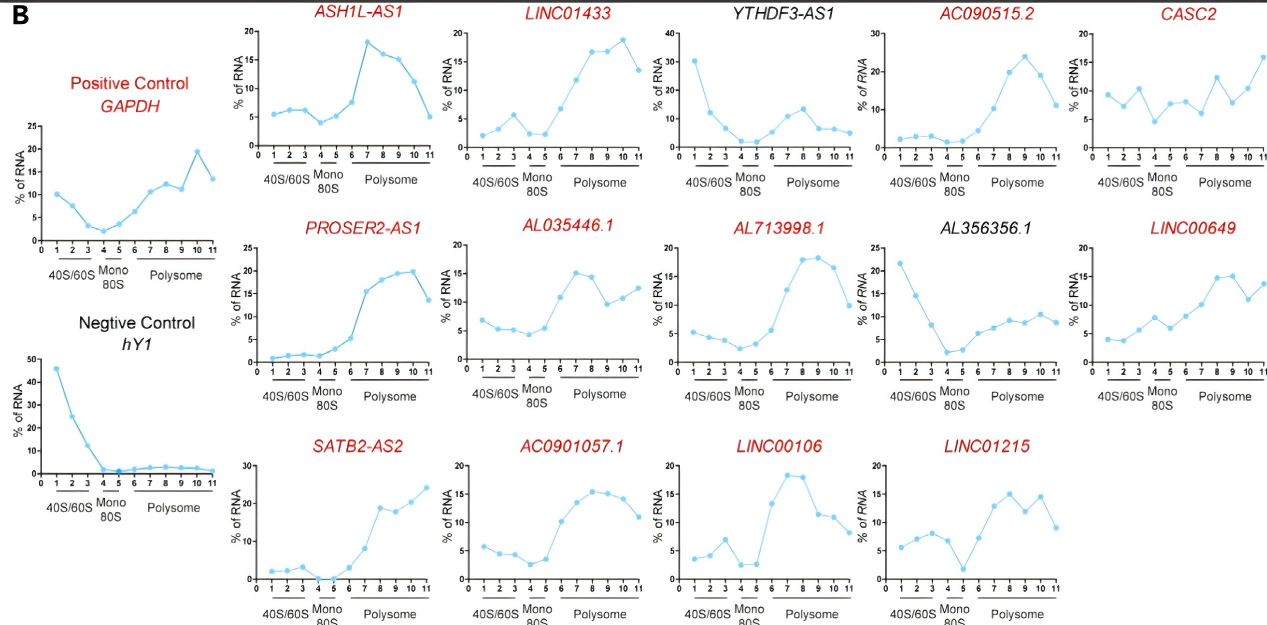
注:A 圖為 Polysome profiling多聚核糖體譜,B 圖為 Polysome-qPCR結(jié)果:目標(biāo)lncRNA的分布。蛋白質(zhì)編碼mRNA GAPDH用作陽(yáng)性對(duì)照。非編碼RNA hY1用作陰性對(duì)照。
【外參法】文獻(xiàn)案例:利用多聚核糖體分析翻譯過(guò)程中mRNA及其相關(guān)蛋白

文章題目:Polysome Profiling Analysis of mRNA and Associated Proteins Engaged in Translation
本方案包括從梯度組分中提取RNA,然后進(jìn)行逆轉(zhuǎn)錄和定量PCR (RT-qPCR)、qPCR數(shù)據(jù)處理。其中引入了熒光素酶RNA(FLuc RNA)做標(biāo)準(zhǔn)化。
qPCR數(shù)據(jù)分析

在所有組分中檢測(cè)到的目的轉(zhuǎn)錄本的總數(shù)設(shè)置為100%,每個(gè)組分中目的轉(zhuǎn)錄本的比例用百分比表示。

4:Polysome profiling+WB
WB/質(zhì)譜驗(yàn)證翻譯活性,還可分析翻譯調(diào)控因子的功能
文獻(xiàn)案例:HectD1通過(guò)調(diào)控核糖體組裝和蛋白質(zhì)合成來(lái)控制造血干細(xì)胞再生

文章題目:HectD1 controls hematopoietic stem cell regeneration by coordinating ribosome assembly and protein synthesis
HectD1 通過(guò)泛素化降解 ZNF622,調(diào)控核糖體組裝和蛋白質(zhì)翻譯效率,從而維持 HSC 在應(yīng)激條件下的再生能力。理論意義:揭示泛素化修飾在核糖體組裝和 HSC 再生中的關(guān)鍵作用,為理解核糖體病的發(fā)病機(jī)制提供了新視角。臨床意義:ZNF622 可能是治療骨髓衰竭綜合征(如 Shwachman-Diamond 綜合征)的潛在靶點(diǎn)。
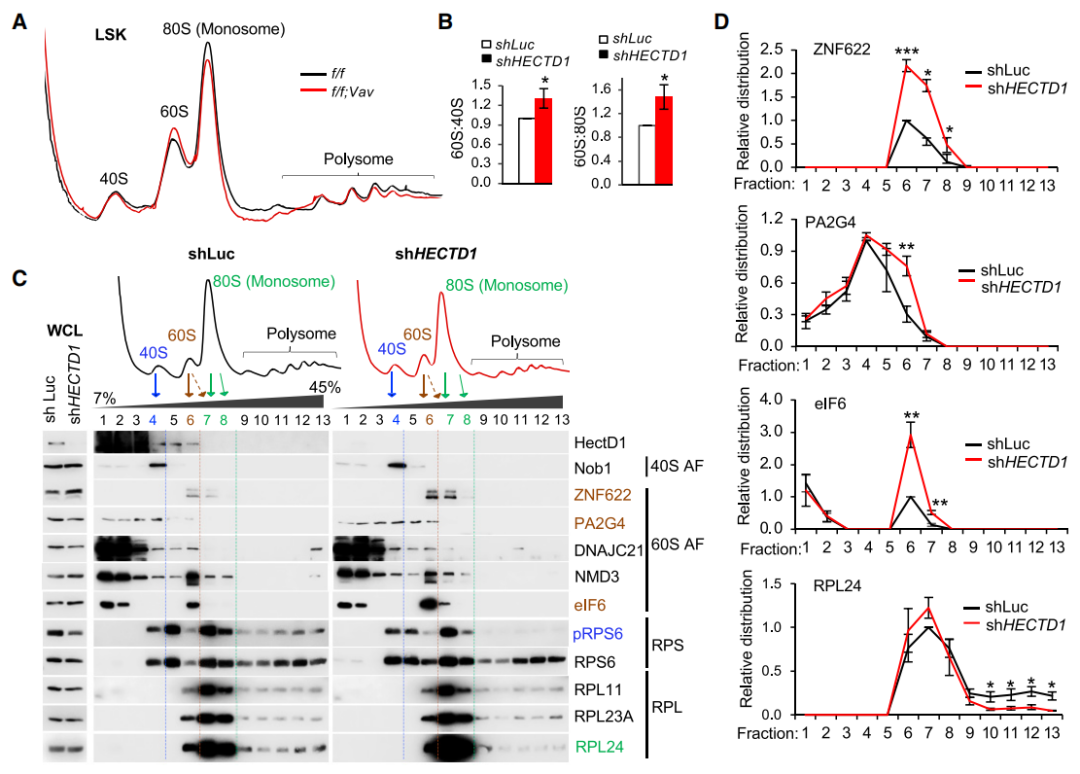
Hectd1 缺失導(dǎo)致 ZNF622 和 eIF6 在 60S 中積累,核糖體亞基連接減少,ZNF622 耗竭后恢復(fù)
注:A、B 圖為 Polysome profiling 峰圖,C、D 圖為 Polysome profiling 的 western blot 分析圖。
參考文獻(xiàn)
1.Eshraghi M, Karunadharma PP, Blin J, Shahani N, Ricci EP, Michel A, Urban NT, Galli N, Sharma M, Ramírez-Jarquín UN, Florescu K, Hernandez J, Subramaniam S. Mutant Huntingtin stalls ribosomes and represses protein synthesis in a cellular model of Huntington disease. Nat Commun. 2021 Mar 5;12(1):1461.
2.Zhao T, Chen YM, Li Y, Wang J, Chen S, Gao N, Qian W. Disome-seq reveals widespread ribosome collisions that promote cotranslational protein folding. Genome Biol. 2021 Jan 5;22(1):16.
3.Jiang F, Hedaya OM, Khor E, Wu J, Auguste M, Yao P. RNA binding protein PRRC2B mediates translation of specific mRNAs and regulates cell cycle progression. Nucleic Acids Res. 2023 Jun 23;51(11):5831-5846.
4.Pringle ES, McCormick C, Cheng Z. Polysome Profiling Analysis of mRNA and Associated Proteins Engaged in Translation. Curr Protoc Mol Biol. 2019 Jan;125(1):e79.
5.Han C, Sun L, Pan Q, Sun Y, Wang W, Chen Y. Polysome profiling followed by quantitative PCR for identifying potential micropeptide encoding long non-coding RNAs in suspension cell lines. STAR Protoc. 2021 Dec 14;3(1):101037.
6.Lv K, Gong C, Antony C, Han X, Ren JG, Donaghy R, Cheng Y, Pellegrino S, Warren AJ, Paralkar VR, Tong W. HectD1 controls hematopoietic stem cell regeneration by coordinating ribosome assembly and protein synthesis. Cell Stem Cell. 2021 Jul 1;28(7):1275-1290.e9.
7.Ryder L, Arendrup FS, Martínez JF, Snieckute G, Pecorari C, Shah RA, Lund AH, Blasius M, Bekker-Jensen S. Nitric oxide-induced ribosome collision activates ribosomal surveillance mechanisms. Cell Death Dis. 2023 Jul 26;14(7):467.
8.Rodriguez-Martinez A, Young-Baird SK. Polysome profiling is an extensible tool for the analysis of bulk protein synthesis, ribosome biogenesis, and the specific steps in translation. Mol Biol Cell. 2025 Apr 1;36(4):mr2.